В декабрьском номере журнала Science, основной темой которого стали орнитологические исследования, вышел цикл статей, освещающий основные результаты работы международного научного коллектива. В его состав вошли более 200 ученых из 20 стран и 80 научных лабораторий. Среди соавторов статей – выпускники НГУ и ученые Денис Ларкин (University of London) и Полина Перельман (Институт молекулярной и клеточной биологии СО РАН).
Группа ученых под руководством Эрика Джарвиса (Duke University) и Годжи Джанга (Beijing Genomics Institute) поставила задачу разрешить филогенетические вопросы с помощью самых современных данных – данных по сравнению ДНК последовательностей всего генома.
До 2008 года учеными был отсеквенирован только один геном птицы – курицы. Потом появились данные по геному земляного вьюрка (2008), индейки (2009), волнистого попугайчика (2011) и древесного вьюрка (2013). Это были первые важные шаги, но существует около 10 тысяч видов птиц. Систематика птиц имеет множество нерешенных вопросов не только на уровне видов-подвидов, но и на высоких уровнях отрядов ученые не могут прийти к консенсусу по поводу порядка ответвления на древе птиц.
Предпринимаемые ранее попытки разрешить спорные вопросы систематики птиц с помощью традиционных методов сравнения по морфологическим признакам или по сравнению ограниченного набора ДНК последовательностей (менее 20 генов) так и не смогли разрешить все вопросы. Идея исследования состояла в том, чтобы отсеквенировав полностью геном и получив данные по генам и районам ДНК, эволюционирующим с различными скоростями, найдя схожие участки ДНК, можно было бы наверняка определить степень родства видов птиц и последовательность их появления в ходе эволюции. Даже полногеномные данные пока не смогли дать на выходе однозначную картину эволюции птиц. Одна из причин – очень быстрое и «одновременное» по меркам эволюции появление множества новых видов птиц, которое произошло после вымирания динозавров, «освободивших» экологические ниши, которые заняли птицы и млекопитающие. Однако многие ответы все же удалось получить.
– Эта статья – новая веха в исследованиях недавно появившегося направления филогеномики – построения эволюционных древ на основе геномных данных, – рассказывает Полина Перельман. – На сегодняшний день отсеквенировано уже немало геномов животных. Однако только для птиц была специально отобрана группа геномов для секвенирования, чтобы потом провести всеобъемлющий анализ всего класса.
Ученые из Новосибирска участвовали в проекте на первых этапах.
– Мы осуществляли проверку принадлежности выделенной ДНК к определенному виду птиц. Например, образец ДНК дальневосточного журавля не был подтвержден и в результате его не выбрали для геномного секвенирования. Нередки случаи, когда при сборе образцов или при их обработке происходит путаница, или существуют трудности в установлении принадлежности к определенному виду или полу, поэтому важно все проверить на нескольких этапах перед секвенированием. Наш отдел разнообразия и эволюции геномов ИМКБ СО РАН является участником проекта “Геном 10К”, возглавляемого Стивеном О’Браейном, Дэвидом Хаслером и Оливером Райдером, который ставит амбициозную цель секвенирования 10 тысяч геномов животных. Серия работ по расшифровке геномов птиц – прямой результат этого исследования, положившего начало новому “Консорциуму Геномов Птиц”, который будет заниматься курированием полученных данных и дальнейшим их изучением”, – отмечает Полина.
– Сейчас наступает один из интереснейших этапов – расшифровки геномных данных. Огромный шаг уже сделан – геномы отсеквенированы, теперь можно приниматься за самые интересные вопросы – исследовать, как работают гены для создания определенного признака (например, внешних признаков, таких как окрас перьев или форма клюва, или признаков, относящийся ко внутреннему устройству, таких как формирование пения, полета, перьев и других характерных для птиц черт). Для нашей лаборатории цитогенетики животных в Институте молекулярной и клеточной биологии интересным и совершенно нерешенным вопросом остается установление связи между данными геномного секвенирования и конкретными носителями генетической информации – хромосомами. А также вопрос преобразования различных наборов хромосом в ходе эволюции.
В ходе же всего проекта по секвенированию и анализу геномов птиц было сделано множество новых открытий, поэтому была опубликована не одна статья, а цикл, в который вошло около тридцати статей.
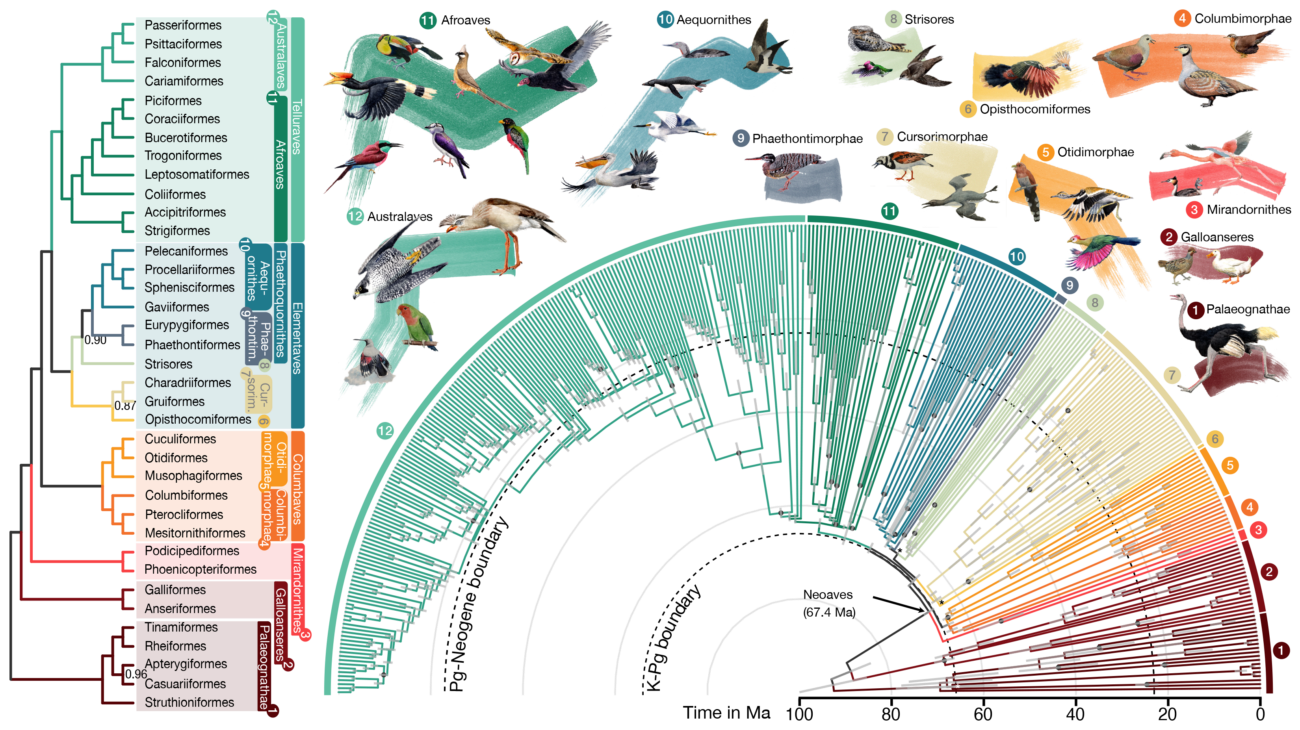
Новое древо птиц из статьи по филогеномике теперь, безусловно, войдет в учебники.
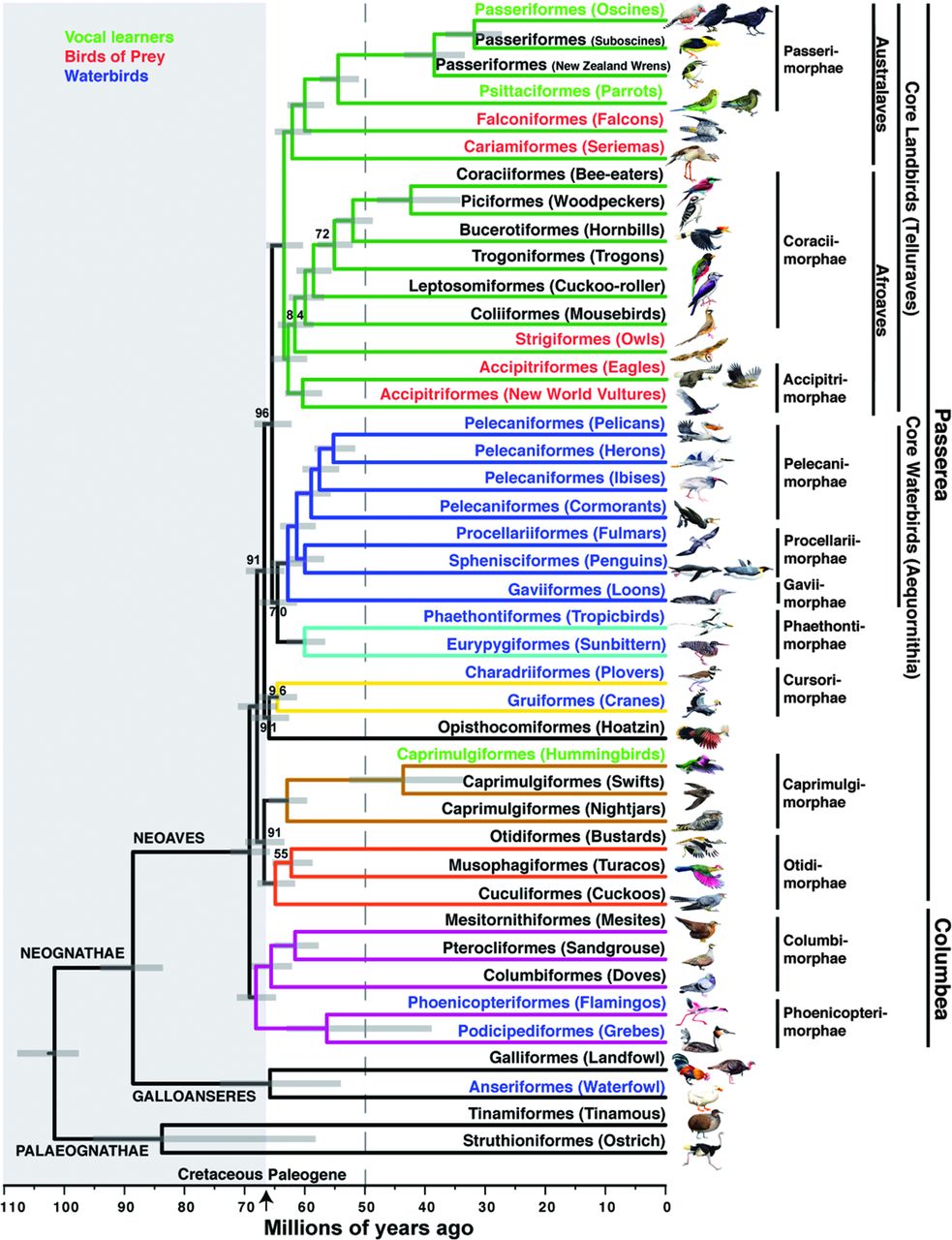
У основания древа птиц отделились нелетающие птицы, потом гусино- и куринообразные. Затем появляется большая группа Neoaves – современных птиц. Здесь ученые выделили две новых систематических группы у птиц – Columbea (фламингообразные, поганкообразные, голубиные, рябкообразные, мадагаскарские пастушки) и Passerea (остальные отряды Neoaves). Когда все отряды были расставлены “по местам”, стало видно, что некоторые морфологические признаки, казалось бы, объединяющие виды в одну группу (способность к вокальному обучению у колибри, попугаев и певчих воробьиных или способность к плаванию под водой у поганок и в другой ветви – у бакланов и гагар), возникали независимо несколько раз в классе птиц.
– Нужно понимать, что древо включает в основном только представителей отрядов, необходимо продолжать работу, чтобы создать детальную систематику на основе данных ДНК для уровней семейств, родов и видов. Биоинформатические инструменты в области анализа сверхпротяженных геномных данных для филогеномных исследований как раз и были созданы в ходе этого проекта. Другой важный вклад проекта – создание базы полногеномных последовательностей основных отрядов птиц. Это необъятный материал для дальнейших исследований, особенно в области сравнения, в том числе и с геномом человека, – заключила Полина Перельман.
Сайт Новосибирского государственного университета 19 декабря 2014 г.
Газета “Наука в Сибири” N50 (2985) от 25 декабря 2014 г.
Подготовила Василиса Петрова

Hiya, I’m really glad I’ve found this info. Today bloggers publish only about gossips and web and this is actually annoying. A good web site with exciting content, this is what I need. Thanks for keeping this site, I will be visiting it. Do you do newsletters? Cant find it.